13 Visualizações Interativas
13.1 Introdução
No R, as visualizações interativas são, geralmente, criadas a partir de pacotes que utilizam um framework chamado htmlwidgets. O hmtlwidgets é um pacote/framework que facilita o uso de bibliotecas javascript de visualizações para o ambiente R, sendo possível usá-las no console, no RMarkdown e no Shiny. Basicamente, javascript é uma linguagem client-side, ou seja, aquelas onde o processamento ocorre no lado do cliente. É utilizada, principalmente, para alterar códigos HTML e CSS (estilos) interativamente.
O htmlwidgets busca fazer a ponte entre o R e alguma biblioteca javascript de visualização, fazendo com que o usuário do R consiga utilizar essas bibliotecas sem necessariamente precisar escrever uma linha de código em javascript. Apesar de não ser necessário, pode ser que, em algum momento, a fim de customizar uma visualização, seja necessário algum código em javascript, mas isto não será tratado aqui.
Abaixo estão listados alguns projetos de visualização de dados em javascript:
Existe uma quantidade significativa de pacotes que utilizam o htmlwidgets. Para se ter uma noção, visite esta galeria.
O objetivo deste capítulo é fazer uma pequena apresentação sobre alguns pacotes. Cada pacote possui um conjunto de detalhes que torna inviável apresentá-los neste curso. Dessa forma, faremos um breve tour por alguns pacotes, começando com o plotly.
13.2 Plotly
É possível ainda dar ainda mais vida aos seus gráficos os transformando em interativos de maneira muito fácil. O pacote plotly, além de ser um ótimo pacote para produzir gráficos interativos em R ou Python, possui uma funcão chamada ggplotly() que transforma um gráfico estático do ggplot2 em interativo.
# criando grafico estatico
library(gapminder)
p <- gapminder %>%
filter(year == 2007) %>%
ggplot(aes(x = gdpPercap, y = lifeExp)) +
geom_point(aes(color = continent))
p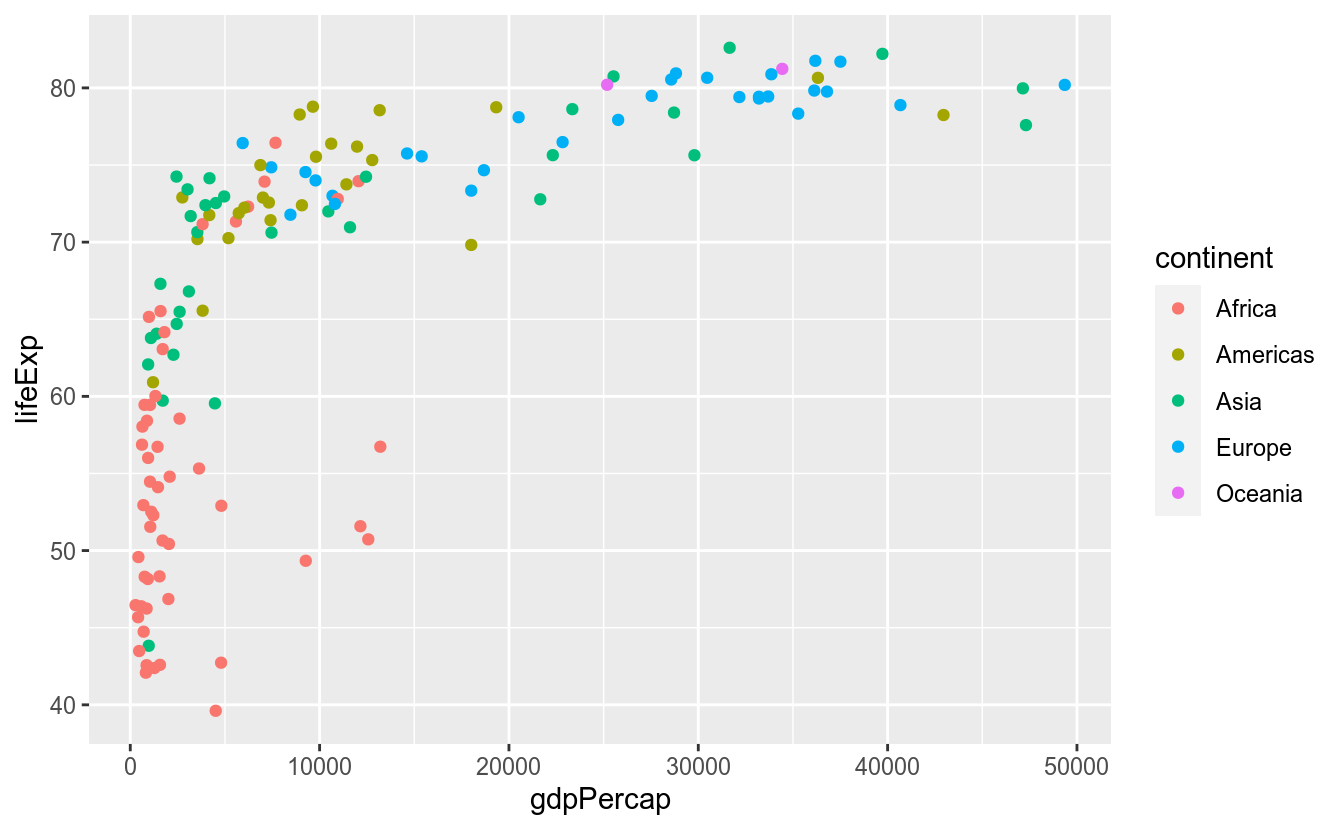
Com apenas uma simples função, temos um gráfico cheio de recursos interativos, como possibilidade de dar zoom em áreas específicos do gráfico e tooltips, que é a pequena tabela de dados que aparece na tela ao passar o mouse em um ponto.
Como era de se esperar, as tooltips também podem ser customizadas. A função ggplotly possui um parâmetro chamado tooltip onde pode ser especificada a aesthetic que será mostrada na tooltip. Por padrão, como você viu, a tooltip mostra todas as aesthetics definidas no gráfico. Caso você queira mudar esse aspecto, pode mudar o valor desse parâmetro em ggplotly:
# mostrar apenas a aesthetic x,
# na qual foi mapeada a variavel de expectativa de vida
ggplotly(p, tooltip = "x")Incrivelmente, dá para ficar ainda melhor. É definindo uma nova aesthetic chamada text, que por padrão não pertence ao ggplot2 mas é usada pelo plotly para construir a tooltip.
Essa nova aesthetic, usada em combinação com a função paste0, pode criar tooltips informativas e elegantes.
Caso não conheça a função paste0(), ela serve para concatenar vetores de strings em um só, de maneira paralelizada.
Segue alguns exemplos:
nome <- c("Lucas", "Eduardo", "Flávio")
sobrenome <- c("Silva", "Oliveira", "Dias")
# concatenar os dois vetores acima, juntando nome e sobrenome com espaço no meio
paste0(nome, " ", sobrenome)## [1] "Lucas Silva" "Eduardo Oliveira" "Flávio Dias"Usando essa função, vamos definir a aesthetic de forma que mostre o nome dos países e os valores das variáveis dos eixos:
# refazer o grafico, definindo uma nova aesthetic chamada text:
p <- gapminder %>%
filter(year == 2007) %>%
ggplot(aes(x = gdpPercap, y = lifeExp)) +
geom_point(aes(
color = continent,
text = paste0("País: ", country, '\n',
"Expectativa de vida: ", round(lifeExp), "\n",
"PIB per capita: ", gdpPercap)
))
ggplotly(p, tooltip = "text")13.3 dygraphs
O dygraphs é uma biblioteca para visualizações de séries temporais. Os detalhes do pacote estão disponíveis neste link.
Antes dos exemplos, será necessário falar sobre objetos de séries de tempo no R.
Para criar um objeto de séries de tempo usaremos a função ts():
ts(data = NA, start = 1, end = numeric(), frequency = 1,
deltat = 1, ts.eps = getOption("ts.eps"), class = , names = )Os parâmetros relevantes são:
data: um vetor ou uma matriz de valores da(s) série(s) de tempo. Um data.frame é transformado automaticamente em uma matriz;start: o período da primeira observação. Pode ser um valor único ou um vetor de dois inteiros. Geralmente, utiliza-se a segunda opção. Por exemplo, Janeiro de 1997:start = c(1997, 1);end: o período da última observação. Similar aostart, porém não é obrigatório;frequency: frequência dos dados. Mensal(12), Trimestral (4), Anual(1) etc.
Exemplo de criação de uma série de tempo:
x <- rnorm(24, mean = 100, sd = 10)
# Trasnformando em série mensal a partir de janeiro de 2010
x <- ts(x, freq = 12, start = c(2010, 1))
plot(x)
Outra maneira de declarar um objeto de série de tempo é utilizando a função xts() do pacote de mesmo nome. No entanto, para essa função, precisamos de um vetor ordenado de datas do tipo Date, POSIXct, timeDate, yearmon e yearqtr.
library(xts)
xts_df <- data.frame(y = rnorm(365, 100, 10))
xts_df$data <- seq.Date(as.Date("2011-01-01"), length.out = 365,
by = "1 day")
xts_df <- xts(x = xts_df[, "y"], order.by = xts_df[, "data"])
head(xts_df)## [,1]
## 2011-01-01 90.87153
## 2011-01-02 100.81501
## 2011-01-03 112.04756
## 2011-01-04 109.55455
## 2011-01-05 101.10612
## 2011-01-06 99.33796library(dygraphs)
lungDeaths <- cbind(mdeaths, fdeaths)
dygraph(lungDeaths,
main = "Mortes por Doenças Pulmonares - Reino Unido - 1874-1979",
ylab = "Número de Morets") %>%
dySeries("mdeaths", color = "blue", label = "Homens") %>%
dySeries("fdeaths", color = "green", label = "Mulheres") %>%
dyRangeSelector()Aqui fica o código para alterar os padrões dos números e datas:
# Alterar rótulos do eixo x e a legenda
axlabform <- "function(date, granularity, opts, dygraph) {
var months = ['Janeiro', 'Fevereiro', 'Março', 'Abril', 'Maio',
'Junho', 'Julho', 'Agosto', 'Setembro', 'Outubro', 'Novembro', 'Dezembro'];
return months[date.getMonth()] + \" \" + date.getFullYear()}"
valueform <- "function(ms) {
var months = ['Janeiro', 'Fevereiro', 'Março', 'Abril', 'Maio',
'Junho', 'Julho', 'Agosto', 'Setembro',
'Outubro', 'Novembro', 'Dezembro'];
var ms = new Date(ms);
return months[ms.getMonth()] + '/' + ms.getFullYear()}"
valueformy <- "function(value) {
return (Math.round(value * 100)/100).toString()
.replace('.', ',')
.replace(/\\B(?=(\\d{3})+(?!\\d))/g, '.')}"
dygraph(lungDeaths,
main = "Mortes por Doenças Pulmonares - Reino Unido - 1874-1979",
ylab = "Número de Morets") %>%
dySeries("mdeaths", color = "blue", label = "Homens") %>%
dySeries("fdeaths", color = "green", label = "Mulheres") %>%
dyAxis("y", valueFormatter = valueformy) %>%
dyAxis("x", axisLabelFormatter = axlabform, valueFormatter = valueform) %>%
dyRangeSelector()13.4 Leaflet
O leaflet é, provavelmente, a principal biblioteca javascript para visualizações interativas de mapas. Mais informações sobre o pacote estão disponíveis neste link. Para iniciarmos uma visualização com leaflet, basta executar o código abaixo. A função addTiles() adiciona uma camada de mapas ao leaflet que foi inicializado.
13.4.1 Primeiro exemplo
No primeiro exemplo, incluiremos um marcador na localização do IBPAD. Para isso, obteremos a latitude e a longitude usando a função geocode() do pacote ggmap. Além disso, foi incluída uma coluna chamada popup, que receberá um texto que será mostrado no mapa.
13.4.2 Marcadores
No exemplo abaixo, criaremos uma visualização com a posição de algumas empresas exportadoras de Mato Grosso, a partir de dados disponibilizados pelo MDIC. Como a busca da localização foi feita usando o endereço, nem sempre a localização estará perfeitamente correta. No entanto, para exemplificar o uso do pacote, não há problemas.
dados.empresas.mt <- read_delim('dados/empresas_exp_mt.csv',
delim = ";",
locale = locale(encoding = 'ISO-8859-1',
decimal_mark = ","))
leaflet(dados.empresas.mt) %>%
addTiles() %>%
addMarkers(lat = ~lat, lng = ~lon, popup = ~EMPRESA)Podemos também adicionar outros tipos de marcadores, como círculos:
leaflet(dados.empresas.mt) %>%
addTiles() %>%
addCircleMarkers(lat = ~lat, lng = ~lon, popup = ~EMPRESA, fillOpacity = 0.3)Adicionalmente, é possível agrupar pontos próximos em clusters.
13.4.3 Polígonos
Também é possível criar-se polígonos a partir de shapefiles.
Para isso, o caminho mais fácil é importar um shapefile usando o pacote sf, criar um gráfico estático com geom_sf() e o converter para interativo com plotly::ggplotly():
# baixa
library(brmap) # remotes::install_github("italocegatta/brmap")
mapa_ufs <- brmap::brmap_estado
head(mapa_ufs)## Simple feature collection with 6 features and 4 fields
## geometry type: POLYGON
## dimension: XY
## bbox: xmin: -73.99045 ymin: -13.6937 xmax: -46.07095 ymax: 5.271841
## CRS: EPSG:4674
## # A tibble: 6 x 5
## estado_cod regiao_cod estado_nome estado_sigla
## <int> <int> <chr> <chr>
## 1 11 1 Rondônia RO
## 2 12 1 Acre AC
## 3 13 1 Amazonas AM
## 4 14 1 Roraima RR
## 5 15 1 Pará PA
## 6 16 1 Amapá AP
## # … with 1 more variable: geometry <POLYGON [°]>## Warning: Ignoring unknown aesthetics: text